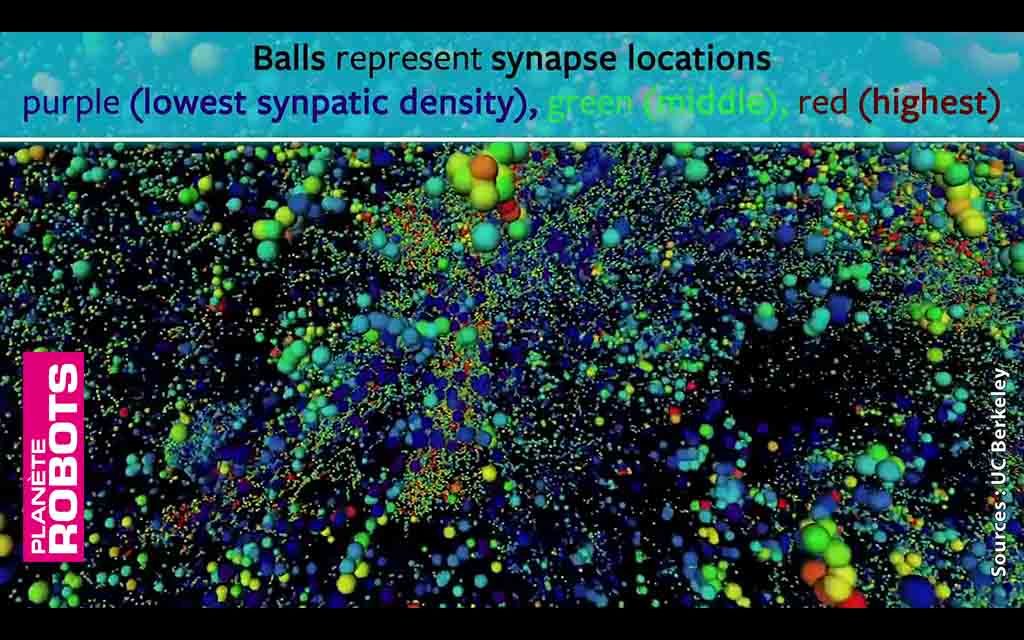
Les chercheurs ont mis au point une nouvelle façon d’imager le cerveau avec une résolution et une rapidité sans précédent. Grâce à cette approche, ils peuvent localiser des neurones individuels, tracer des connexions entre eux et visualiser les organites à l’intérieur des neurones, sur de grands volumes de tissu cérébral.
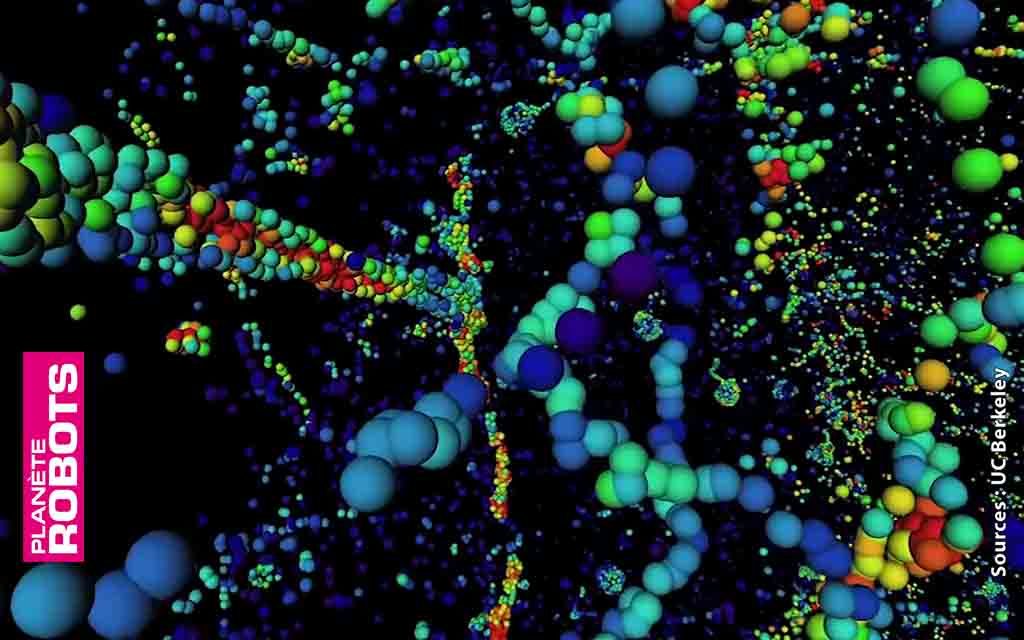
Cette nouvelle technologie combine une méthode d’expansion des tissus cérébraux, qui permet d’obtenir des images à plus haute résolution, avec une technique de microscopie 3D rapide connue sous le nom de microscopie en réseau à feuille légère. Dans un article paru dans Science le 17 janvier, les chercheurs ont montré qu’ils pouvaient utiliser ces techniques pour visualiser le cerveau entier de la mouche des fruits, ainsi que de grandes sections du cerveau de la souris, beaucoup plus rapidement qu’auparavant. L’équipe comprend des chercheurs du MIT, de l’Université de Californie à Berkeley, du Howard Hughes Medical Institute et de la Harvard Medical School/Boston Children’s Hospital.
En 2015, le laboratoire de Boyden a mis au point un moyen de générer des images à très haute résolution des tissus cérébraux à l’aide d’un microscope optique ordinaire. Leur technique repose sur l’expansion des tissus avant l’imagerie, ce qui leur permet d’obtenir des images du tissu à une résolution d’environ 60 nanomètres. Auparavant, ce type d’imagerie ne pouvait être réalisé qu’avec des microscopes à haute résolution très coûteux, connus sous le nom de microscopes super-résolution.
Dans la nouvelle étude, Boyden s’est associé à Betzig et à ses collègues du campus de recherche Janelia de l’IHMH pour combiner la microscopie d’expansion avec la microscopie à réseau optique. Cette technologie, que Betzig a développée il y a plusieurs années, possède des caractéristiques clés qui la rendent idéale pour être couplée à la microscopie à expansion : Il permet d’obtenir rapidement des images de grands échantillons et induit beaucoup moins de lésions que d’autres techniques de microscopie fluorescente.
L’imagerie d’échantillons de tissus élargis génère d’énormes quantités de données – jusqu’à des dizaines de téraoctets par échantillon – de sorte que les chercheurs ont également dû concevoir des techniques de traitement d’images informatiques hautement parallélisées qui pourraient décomposer les données en petits morceaux, les analyser et les recoudre en un ensemble cohérent.
Dans ses travaux futurs, Boyden envisage d’utiliser cette technique pour tracer les circuits qui contrôlent la formation et le rappel de la mémoire, pour étudier comment les entrées sensorielles conduisent à un comportement spécifique, ou pour analyser comment les émotions sont couplées à la prise de décision…
sources :
https://www.media.mit.edu/articles/mapping-the-brain-at-high-resolution/



